MR Cole Photographer/Getty Images
În 2003, genomul uman a fost secvențiat „complet”. În 2022, s-a întâmplat iar. Stai, ce?26 min read
Tabloul complet al genomului uman este (aproape) finalizat la două decenii de la Human Genome Project
Printre știrile despre război sau alegerile controversate din alte state s-a strecurat, la începutul lunii aprilie, și o informație mai optimistă: genomul uman a fost secvențiat complet. Numai că, deși sună ca o bornă majoră în istoria științei (și chiar este), informația ar putea să te lase confuz dacă erai la curent cu noutățile de pe la începutul anilor 2000. Nu fusese genomul completat în primii ani ai acestui mileniu, în urma uneia dintre cele mai mari colaborări științifice din istorie, al cărei succes a fost chiar „Clinton and Blair hail gene ‘triumph’”, theguardian.com
Răspunsul este puțin mai complicat și nu, nu include efectul Mandela. Într-adevăr, genomul a fost anunțat ca fiind secvențial complet la acea vreme. Și nu doar în discursuri exagerate ale politicienilor care voiau să se identifice cu o parte din succes, ci și de comunitatea științifică.
Varianta simplă este că, atunci, cercetătorii implicați au secvențiat tot ce puteau secvenția cu tehnologia pe care o aveau, care se nimereau să fie și părțile de care erau cel mai mult interesați.
Acum, cercetători dintr-o nouă colaborare internațională de amploare, Consorțiul Telomere 2 Telomere (T2T) – numit după telomeri, părțile de la capetele cromozomilor în care se află mare parte din genomul rămas nedescifrat și a căror deteriorare „Telomeres in cancer: tumour suppression and genome instability”, nature.com – susțin că au reușit, de data aceasta, să secvențieze chiar întreg genomul uman.
Pentru varianta puțin mai complicată, care explică ce exact au reușit să completeze cercetătorii acum și de ce aceste părți nu au putut fi descifrate de Human Genome Project, trebuie mai întâi trecut printr-o explicație scurtă a genomului.
Ce este genomul?
Cele mai multe manuale definesc genomul ca totalitatea informației genetice a unui organism. Este destul de ușor de confundat genomul cu termenul de la care îi vine numele, genele – „DNA”, genomenewsnetwork.com Însă genomul nu e doar o colecție de gene.
Genele sunt, într-adevăr, parte a genomului, însă una relativ mică: doar aproximativ 2% din genomul uman este alcătuit din gene care codează proteine. Restul de 98% este reprezentat de alte elemente, care ajută mai mult sau mai puțin alte tipuri de procese biologice (precum transcrierea informației în ARN), de exemplu secvențe repetitive sau copii ale genelor.
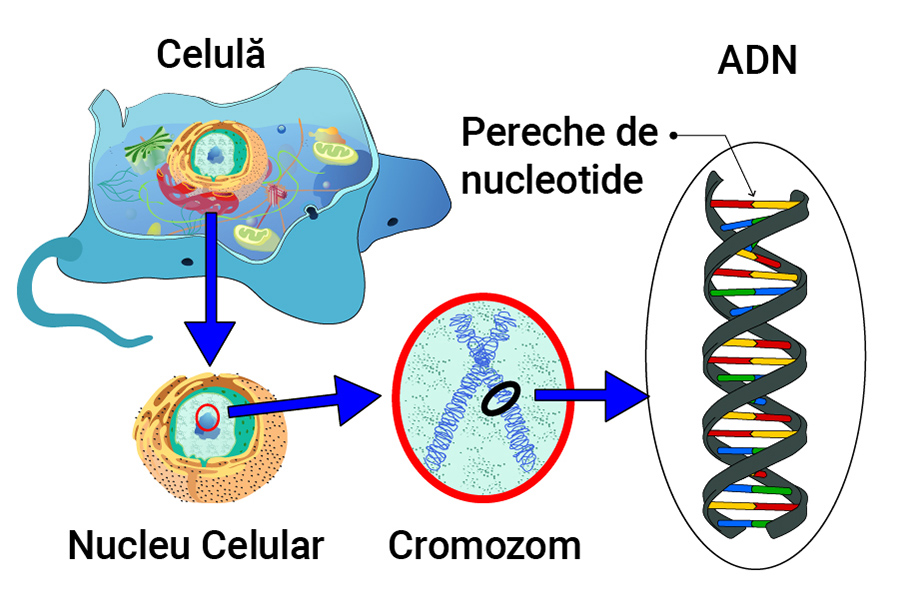
Cum se diferențiază aceste structuri depinde de modul în care este alcătuit și ordonat ADN-ul sau, mai specific, elementele sale de bază, molecule organice numite nucleotide. În structura arhicunoscută a ADN-ului, de spirală dublă, nucleotidele formează perechi de baze („liniile” care unesc elicele) din patru compuși organici, adenină-timină și guanină-citozină.
Genomul uman conține peste trei miliarde de astfel de perechi de nucleotide. Secvențe din AND formate din mai multe nucleotide pot avea roluri diferite, de la cel de codificare (pe care le definim drept gene) la unele structurale și sunt, destul de des, repetitive. . În celule, ADN-ul se strânge în molecule lungi, numite cromozomi; cea mai mare parte a oamenilor au 22 de perechi de cromozomi (numiți autozomi) și O pereche de cromozomi X determină sexul feminin al fătului, în timp o pereche X+Y determină sexul masculin Aceștia conțin, practic, întregul genom.
Secvențierea genetică observă ordinea perechilor de nucleotide și a segmentelor repetitive mai largi pe care le formează în fiecare cromozom și face o „hartă” a acestora. Aceasta hartă genomică este una de referință, fără variațiile genetice individuale care contribuie la diversitatea speciei umane (sau a mutațiilor care pot crește riscul de boli grave), însă poate releva fragmentele specifice din genomul nostru în care apar aceste variații.
Wikimedia Commons
De ce nu a fost completat genomul în anii 2000?
Proiectul original de mapare a genomului uman, Human Genome Project, a fost „What is the Human Genome Project?”, genome.gov În momentul în care a început, în 1990, programul și-a propus să secvențieze întreg genomul uman în 15 ani. A avut nevoie de mai puțin decât atât, chiar dacă succesul anunțat de politicieni în 2000 a fost puțin prematur. La acel moment a fost secvențiat doar un draft al genomului uman. Practic, cercetătorii obținuseră o secvențiere de referință care trebuia verificată pentru a reduce erorile la un nivel minimal înainte de a fi omologată.
Această verificare trebuia să ducă acuratețea secvențierii la 99,9% (mai puțin de o eroare la 10.000 de perechi de nucleotide) și să mapeze toate regiunile care puteau fi analizate cu ajutorul tehnologiei disponibile atunci. Verificarea „International Consortium Completes Human Genome Project”, genome.gov iar secvențierea genomului a fost omologată oficial în 2004.
Comunicarea de la acea vreme este vinovată de confuzia asupra „completării” genomului uman. Anunțul verificării a fost însoțit de afirmația atent selectată că secvențierea este „esențialmente completă”. Nu e neapărat o afirmație falsă, fiindcă se bazează pe două argumente solide: au fost secvențiate toate zonele posibile cu tehnologia de la acea vreme, iar harta obținută avea 99% din regiunile care conțin genele propriu-zise, considerate mai importante decât orice alt tip de ADN.
Per total, însă, în jur de 8% din codul genetic uman „ The Human Genome Project pieced together only 92% of the DNA – now scientists have finally filled in the remaining 8%”, theconversation.com Fix aceasta este bucata care a stat la baza unei alte colaborări internaționale, Consorțiul Telomere 2 Telomere (T2T), care a strâns zeci de cercetători din SUA, Marea Britanie, Germania și chiar Rusia pentru a oferi o variantă chiar completă a genomului.
Doar că și aici există o problemă de nuanță a limbajului – chiar dacă informația a fost publicată din nou „The complete sequence of a human genome”, science.org Science, jurnalul care a publicat articolul principal al cercetărilor T2T, l-a prezentat drept „Most complete human genome yet reveals previously indecipherable DNA”, science.org Diferențele, în ambele cazuri, sunt mai degrabă tehnice și pot fi observate, cel mai ușor, în unele particularități ale genomului uman.
Nu tot ADN-ul este împachetat la fel
Există o diferență majoră între genomul secvențiat prin Human Genome Project și cel abordat de T2T: „What is chromatin, heterochromatin and euchromatin?”, mechanobio.info
Unitatea de măsură în acest sens este cromatina – o substanță alcătuită din bucăți de ADN, ARN și anumite proteine, care creează structuri compacte din moleculele lungi de ADN. Asta previne încrucișarea moleculelor și le face mai solide, pentru a evita deteriorarea ADN-ului atunci când celule noi se formează prin diviziunea celor deja existente. În funcție de forma în care sunt împachetate aceste molecule, există două tipuri cromatină în genomul uman.
Cea mai răspândită este eucromatina, care formează unități repetitive de cromatină cu aspect similar unui șir de mărgele pe ață. Acesta este varianta mai puțin compactă de împachetare și este folosită în 92% din genomul uman – cel care a fost secvențiat prin Human Genome Project.
A doua formă este heterocromatina, care împachetează ADN-ul în structuri mult mai dense și conține și o proporție mult mai mare de ADN repetitiv, precum și semnificativ mai puține regiuni cu gene decât eucromatina. Din aceste motive, i s-a dat și supranumele formal de „junk DNA”. Heterocromatina este prezentă, de obicei, În reprezentările cunoscute sub formă de X ale cromozomilor, centromerii ar fi punctele în care se intersectează cele două șiruri de cromatină și la capetele cromozomilor (telomeri).
Cu tehnologiile folosite în cadrul Human Genome Project, care puteau citi „DNA Sequencing Technologies Key to the Human Genome Project”, nature.com se putea stabili secvența exactă a ADN-ului eucromatic pe baza șirurilor de nucleotide care se suprapuneau, oarecum similar unui puzzle gigantic. Această capacitate era, însă, insuficientă pentru secvențierea fragmentelor mult mai dense de heterocromatină, care necesita identificarea simultană a unor blocuri mult mai largi pentru a putea găsi șirurile care se suprapun.
În plus, la momentul respectiv încă se bănuia că o parte semnificativă din fragmentele repetitive de ADN, în special cele grupate foarte dens, precum heterocromatina, nu jucau un rol biologic util sau chiar activ, fiind secvențe pasive acumulate de-a lungul evoluției genetice umane. Între timp, această ipoteză s-a modificat, treptat, odată cu apariția „The Complex Truth About ‘Junk DNA’”, quantamagazine.org despre rolul pe care diferitele tipuri de ADN-uri ne-codant îl joacă în procesele biologice. Asta a dus la o mișcare științifică prin care se încearcă retragerea termenului de junk DNA.
Se poate înțelege, însă, de ce la acea vreme genomul era considerat „esențialmente complet”, atunci când barierele actuale până la completarea acestuia erau, din perspectiva cercetătorilor, imposibil și inutil de secvențiat.
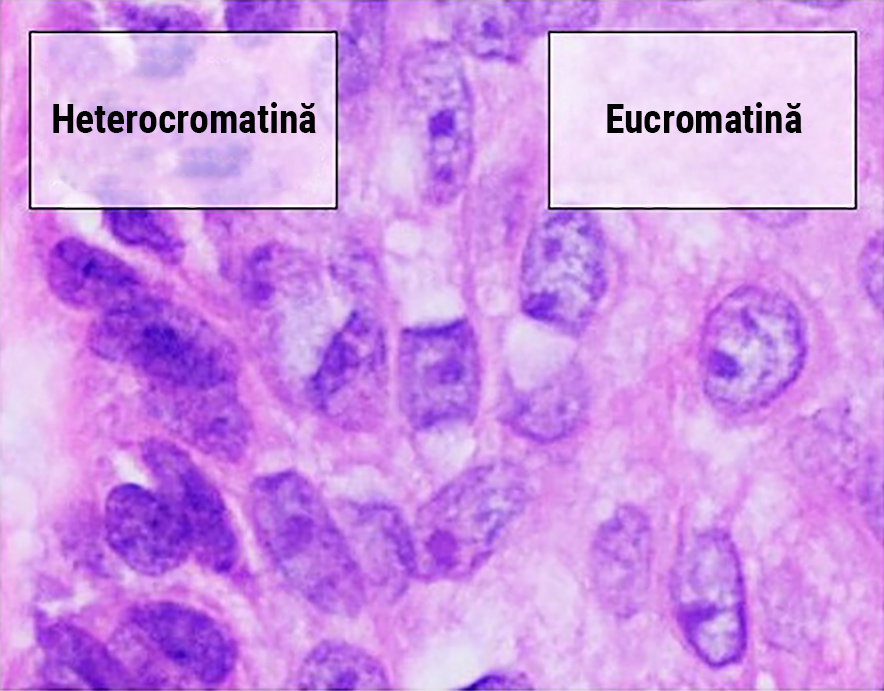
Diferențele microscopice dintre heterocromatină și eucromatină. ADN-ul heterocromatic este grupat mult mai dens, având o culoare mai închisă. Mikael Haggstrom/Wikimedia Commons
Cum a fost completat genomul uman?
Chiar dacă se estima că dezvoltarea tehnologiilor necesare pentru a secvenția regiunile lipsă ale genomului ar putea dura câteva decenii, acestea „Filling in the gaps telomere to telomere”, nature.com Consorțiul T2T a folosit trei tehnologii recente de secvențiere care, în studii publicate între 2015 și 2019, au demonstrat potențialul de a citi fragmente lungi, de până la 100.000 de perechi de nucleotide simultan, cu acuratețe ridicată; două au fost produse de compania americană Pacific Biosciences și una de britanicii de la Oxford Nanopore Technologies.
În urma mai multor teste efectuate pe diferite genomuri parțial descifrate, cercetătorii T2T au selectat și linia genomică care avea șansele cele mai ridicate de secvențiere completă. Numită CHM13, ea nu provine direct de la un donator uman, ci a fost extrasă de la o complicație rară care poate apărea în anumite sarcini, numită „Molar pregnancy”, wikipedia.org
În cadrul acesteia, un ovul non-viabil, care nu are un nucleu matern, este fertilizat cu succes, creând un țesut al cărui genom are perechi de cromozomi identici de la tată – spre deosebire de un genom uman normal, unde perechile de cromozomi provin de la ambii părinți. Asta reduce semnificativ dificultatea secvențierii, deoarece cercetătorii nu mai trebuie să descifreze două seturi diferite de cromozomi.
Tehnologiile au fost folosite inițial pentru a secvenția cromozomul X din linia CHM13, iar rezultatele au fost puse cap-la-cap pentru a alcătui o hartă de referință a acestuia. Secvența obținută avea doar trei breșe, care au fost rezolvate de cercetători printr-o combinație de identificare manuală a secvențelor lipsă și folosirea de algoritmi construiți special pentru „centroFlye: Assembling Centromeres with Long Error-Prone Reads”, researchgate.net și „TandemTools: mapping long reads and assessing/improving assembly quality in extra-long tandem repeats”, oup.com
Astfel, a fost obținută o secvență completă a cromozomului X, cu acuratețe de peste 99,9%. Metoda a fost folosită, apoi, cu succes și pentru secvențierea completă a unui autozom, cromozomul 8, ceea ce a deschis posibilitatea unei secvențieri a complete a întreg genomului liniei CHM13. Aceasta a fost anunțată, inițial, în septembrie 2020, iar după completări și verificări prin peer review, a fost publicată oficial la sfârșitul lui martie ca „The complete sequence of a human genome”, science.org
„Complet” nu înseamnă neapărat complet
Totuși, la fel ca și declarația genomului „esențialmente complet”, și acest anunț ascunde câteva nuanțe care pun în discuție ideea unui genom uman secvențiat integral.
Linia genomică publicată, sub numele Disponibilă pe github.com adaugă peste 200 de milioane de perechi de nucleotide, incluzând 99 de gene care pot coda proteine. Din secvențierea inițială lipsea, însă, cromozomul Y, deoarece linia CHM13 folosită provenea de la o sarcină molară fecundată cu cromozomul X. Între timp, acesta a fost secvențiat dintr-o altă linie donată de un biolog de la Harvard și adăugat genomului de referință, iar cercetătorii T2T au anunțat că va urma și un studiu care va detalia acest aspect.
Sunt, însă, cinci zone din genomul T2T, cu aproximativ 10 milioane de perechi de nucleotide, care nu au putut analizate în detaliu. Mai exact, în aceste zone nu a putut fi determinată și verificată ordinea exactă a perechilor, așa că secvențierea lor se bazează mai mult pe estimări.
Așa că desemnarea liniei T2T-CHM13 drept una completă ține mai mult de tehnicalități – este într-adevăr primul genom uman publicat fără breșe, însă s-ar putea puncta la fel de bine faptul că acesta nu va deveni complet până la omologarea prin peer-review a secvențierii cromozomului Y și rezolvarea celor cinci regiuni problemă.
Dincolo de terminologie, ce s-a aflat mai exact din regiunile nou-secvențiate? Atribuirea unor efecte sau ipoteze clare ale acestora asupra proceselor biologice umane poate fi făcută doar prin studii ulterioare, însă structura ADN-ului proaspăt catalogat poate oferi niște indicii.
De exemplu, deși se bănuiește că fragmentele din centromere joacă același rol în toți cromozomii, acela de a-i întări structural, secvențele repetitive în sine sunt de mărimi diferite, invitând o analiză asupra unor posibile altor funcții ale acestora. Pe de altă parte, mutațiile sau modificările chimice ale fragmentelor de ADN repetitiv complexe ar putea fi legate de unele boli genetice, având în vedere că s-au propus în trecut legături între unele afecțiuni neurologice și tulburări de dezvoltare și variații din copiile unor secvențe repetitive specifice.
Astfel de indicii vor putea fi cercetate doar în urma secvențierii unui număr cât mai ridicat și diferit de genomuri umane, iar cercetătorii de la T2T se mișcă deja înspre acest scop. De data aceasta ca parte a Mai multe detalii pe humanpangenome.org ei își propun să secvențieze linii genetice de la 350 de persoane de diverse origini, iar munca la 70 dintre acestea deja a început.
CITEȘTE ȘI: 8 boli care ar putea fi tratate cu ajutorul editării genetice CRISPR





